作者:唐骋(中国科学院脑科学与智能技术卓越创新中心)
文章转载自中国科普博览大家族科学大院(ID:kexuedayuan)
了解演化的朋友应该都知道,只要往上追溯,任何两个生物都能追溯到某个共同祖先,而祖先还能继续溯源到祖先的共同祖先,最终构建起一棵足以囊括古今众生的演化之树。
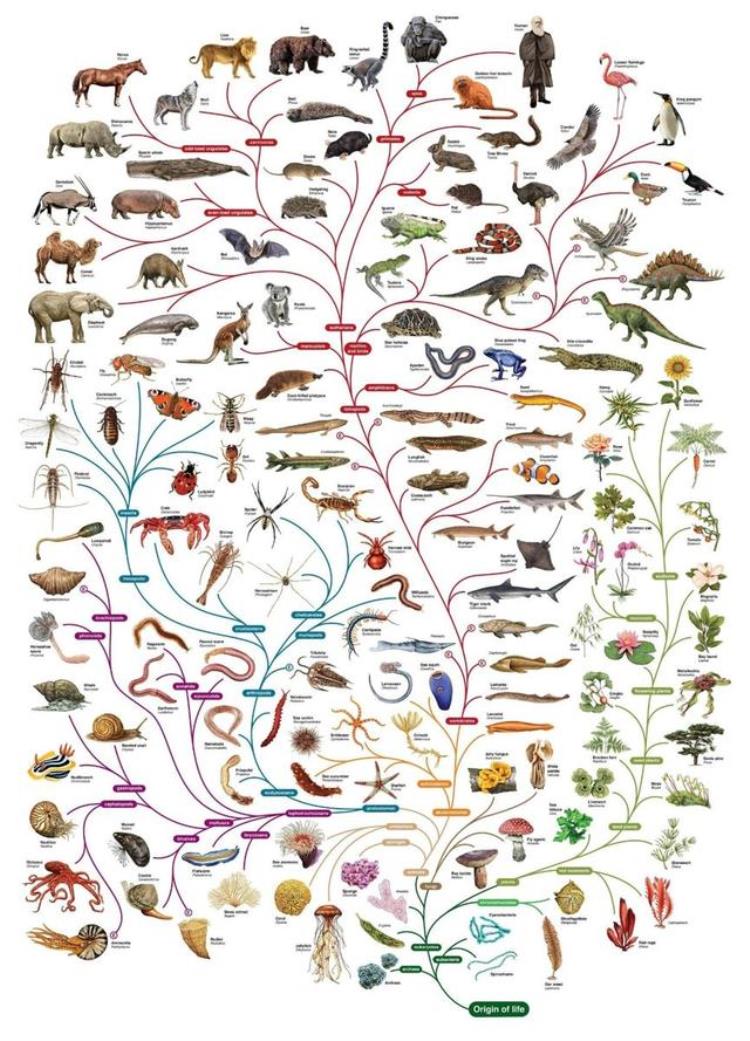
大家熟悉的演化树大概就是这个样子的(图片来源:Pinterest;上传者:Michelle West)
然而病毒却似乎不在这棵树上。大部分病毒的结构很简单,基本上就是一个蛋白质构成的外壳包着一些遗传物质,平时就与死物无异,一旦感染细胞,它们就将自己的基因注入宿主细胞当中,利用宿主细胞复制自身产生新的病毒。这种奇怪的生活方式让我们难以将其置于演化之树的任何地方。
病毒是生物吗?它们又从何而来呢?
下面,我们就来看看病毒起源的三个假说。
假说一:远古分子生命的复仇之魂病毒的故事或许还要从远古说起。在那个了无生机的地球上,海洋占据了世界的绝大部分,而无垠的大海之下是数亿年连绵不断的火山活动。在某些机缘巧合之下,海水中产生了史上第一批有机大分子,从而点燃了生命的星星之火。

生命最初或许就诞生在类似这样的地方,Credit: Ocean Exploration Trust
慢慢的,有一些大分子,比如说RNA,DNA以及蛋白质开始或独立或彼此协作地复制自身,这个阶段可以称为“分子生命”。然而在一片祥和的分子生命中却产生了一个异类,那就是包括现存所有生物的最近共同祖先“露卡”(LUCA)在内的一些分子生命,它们发展出了一个改变了生命法则的结构,那就是细胞。拥有膜结构的细胞可以更好地保护其中娇弱的RNA和蛋白质等核心大分子,极大增强了这些生物的适应力,意味着它们会把原始的分子生命摁在地上摩擦。
而有一种假说(The Virus-First Hypothesis)就认为,病毒正是原始分子生命世界的遗民,它们在在最后一刻创造出了寄生的生活方式,反过来利用了敌人的细胞结构,宛如一群远古的复仇之魂,对于夺取它家园的族裔作着永恒的抗争。
这个假说一度十分盛行,毕竟病毒的构造是如此简单,乃至简陋,它们与细胞生命的差异又是如此巨大。
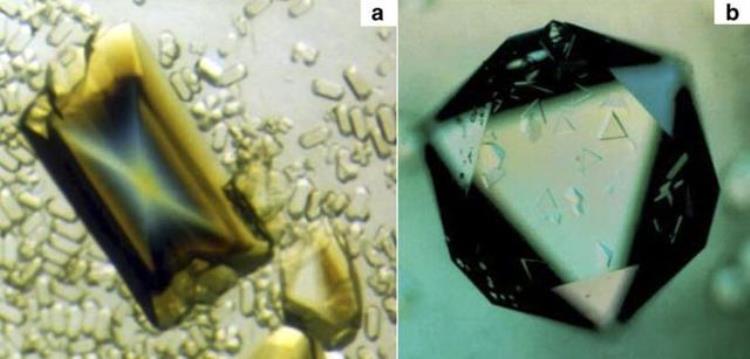
△有些病毒经提纯后还能形成结晶,这对于细胞生物是无法想象的(图片来源:Alexander McPherson & Lawrence James DeLucas)
更实锤的证据来自于“拟病毒”(Virusoid),它就是一个RNA分子。拟病毒没办法直接感染细胞,但是它可以感染病毒,确切地说是在某些病毒感染细胞的时候通过搭便车来顺便复制自己、扩散自己,从而能引发一些诸如人类丁型肝炎之类的疾病。
然而这个热血的病毒起源故事却随着分子生物研究学的深入而显得不再那么完美……
假说二:“叛逃”的基因基因这浓眉大眼的也能叛变?对,在这个假说(The Progressive Hypothesis)里,基因为了让自己流传下去什么事情都做得出来。
细菌中广泛存在一种名叫“质粒”(plasmid)的小片段环状DNA,这些基因基本上就是一群打工仔临时工,细菌随时可以从环境中吸收它们为己所用,也随时可以赶走它们。

△在很多细菌中,除了自身原本的DNA(红色部分)以外,还经常会有一些小片段的环状DNA(橙色),那些被称为质粒(图片来源:作者绘制)
于是在漫长的演化中,有些质粒学会了一件事:我们不要一辈子打工!这些质粒从打工仔变成了二五仔,反过来把它们的细菌老板给劫持了,夺走了细菌所有的营养来复制自身。随着时间推移,有些质粒就变成了病毒。
△细菌经常会遭受一类叫做“噬菌体”(Phage)的病毒感染,有些观点认为噬菌体来源于质粒(图片来源:Quanta Magazine)
而我们人类以及所有动物、植物的细胞和细菌很不一样,我们都属于“真核生物”,细胞当中并没有细菌那样的质粒,但还是有那么一些基因蠢蠢欲动。它们不肯在染色体上好好待着,而是在细胞核里左右横跳,一会儿跑到这个染色体上,一会儿跑到那个染色体上。不过这帮调皮捣蛋的基因倒是有一个仙风道骨的名字——转座子(transposon)。
根据分子生物学检测,很多转座子都拥有和病毒非常相似的基因序列,两者将自己整合进宿主细胞染色体的机制也高度相似。尤其是其中的“病毒样逆转录转座子”(Retrotransposon)与某些病毒简直相似到了令人发指的程度,唯一的不同只是这些转座子还不能像病毒一样在细胞之间迁移而已。
然而病毒的身世到这里却依旧扑朔迷离,因为它们根本就不遵循一般生物的演化模式。
病毒感染的一般流程是:把自己的基因注入宿主细胞当中,然后利用宿主细胞复制自己的基因,并且制造构建病毒颗粒所需的各种材料,最后操控细胞将病毒颗粒的各个零件连同病毒的基因一起包装成新的病毒颗粒,释放出去感染别的细胞。
但是在这个过程中,病毒每时每刻都在和各种生物交换基因。
比如说病毒在指挥宿主细胞包装病毒颗粒的时候,有时候会把一些宿主细胞的DNA给一块包进去,或者不小心在宿主细胞当中留一点点自己的基因。哺乳动物中有一个用来阻止母体免疫系统攻击胎儿的基因,就是某个病毒在一亿多年不小心落在我们细胞中的。而在病毒跨物种传播的时候,经常导致基因从一个物种转移到另一个物种,所以,“转基因”这件事都是自然界玩剩下的。
然而也正是因为病毒的转基因如此之频繁,让人不得不重新思考,病毒真的是叛逃的基因吗?我们会不会从一开始就搞错了逻辑关系。那些转座子啥的,也许并不是不安其室的自身基因,反而是偶然留在细胞中的病毒基因呢?
假说三:病毒也许是沉沦的细胞生物进入21世纪以后,一系列发现开始让科学家愈发意识到病毒的起源还有别的可能性。
2003年,科学家发现了一种非常不讲道理的病毒——“拟菌病毒”(Mimivirus),这个病毒的体型达到了0.4到0.5微米,在显微镜下看都快跟细菌差不多了。引发这次疫情的冠状病毒都被认为是比较大的一类病毒了,体型也不到0.1微米。
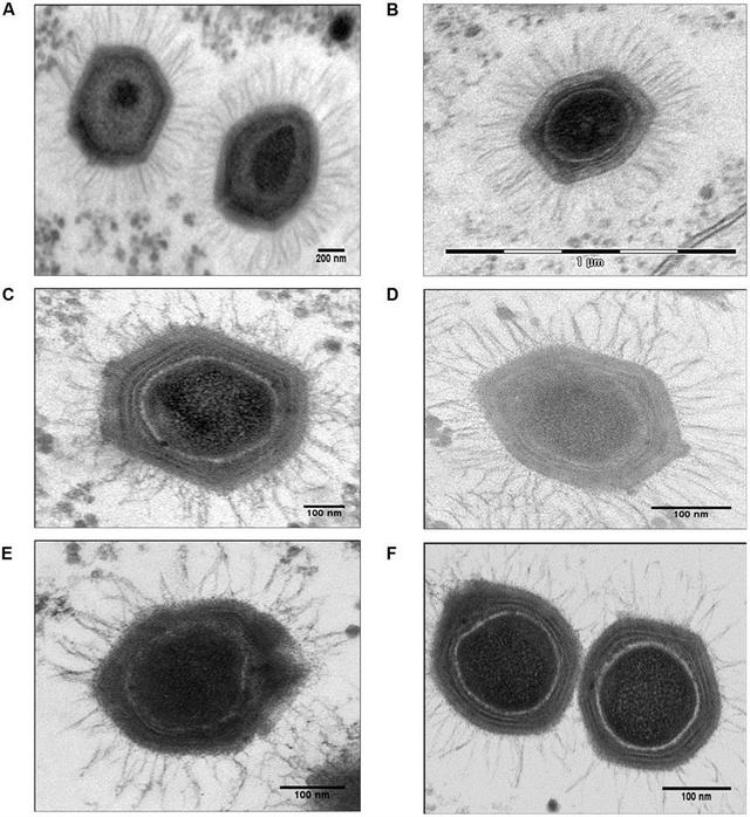
庞大的拟菌病毒(图片来源:Haitham Sobhy et al. 2015)
2008年,科学家发现了第二种大病毒,将其命名为 “妈妈病毒”(Mamavirus),从此一种又一种“大病毒”(Giant Virus)开始接二连三地出现在人类的视野中,到2013年发现的“潘多拉病毒”(Pandoravirus)更是把最大病毒的记录刷到了1微米以上。
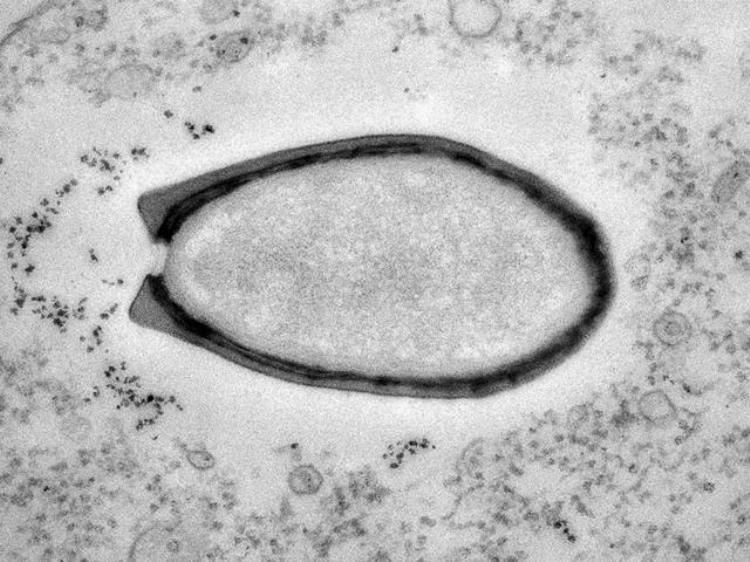
△目前已知最大的病毒“潘多拉病毒”,它看上去和一个细菌几乎没什么区别(图片来源:美国国家地理;作者:Chantal Abergel和Jean-Michel Claverie)
从此,病毒与某些单细胞生物的界限开始变得模糊了起来,比如说“拟菌病毒”的结构与基因和一类叫做“古菌”的单细胞生物非常相似,唯一的不同是,拟菌病毒丢失了一部分自主完成细胞分裂的关键基因,所以不得不寄生在其它生物的细胞当中,利用宿主的细胞来生长繁殖。
于是就有了病毒起源的第三种假说(The Regressive Hypothesis),认为病毒本质上是堕落的生物。有些单细胞生物在长期寄生生活中,逐渐退化掉绝大部分细胞结构,最终变成了这种活死人一般的样子,而像拟菌病毒之流就是刚刚开始堕落的古菌。
在我们的演化中,病毒留下了深深的烙印那么,病毒究竟应该是远古的复仇之魂,还是叛逃的基因,亦或是堕落的生物呢?这三种假说都有合理之处,同时也存在着不能解释的问题。也许,病毒的起源故事远比这一切假说都更加复杂,甚至未必有着唯一的来源。
我们可能永远也弄不清病毒从何而来,但是病毒却真真切切地在我们的演化中留下了深深的烙印。
由于病毒无比强大的变异能力,我们永远也无法预测何时何地会出现一个对我们有致命威胁的病毒,最终逼得很多生物不得不采取一些用变异打败变异的手段。
比如说有性生殖。有性生殖带来最直接的后果就是可以让我们的每一个后代的基因都能打乱重排,尽可能保障所有后代不至于被一种病毒一锅端了。病毒与生物的军备竞赛也一直是演化的最强推手,病毒还打破了生殖隔离的壁障,让基因得以在不同的生物中流动。
所以,病毒虽不属于演化之树上的任何一个枝丫,但它们宛若是围绕在演化之树上的流萤与鬼魅,在这棵树上处处都留下了自己的痕迹。
那么为什么致命病毒总是来自于野生动物呢?让我们从演化的角度分析一下。
“最好”的病毒,是既不引起太严重的症状(不然搞死了宿主自己也完蛋),但也不能太温和(毕竟宿主身上往往同时寄生着别的病毒,抢资源的时候该下的狠手还是得下)。所以在漫长的演化中,这种博弈会促使病毒最终与宿主达成某种默契,比如说人类与可能会引起普通感冒的鼻病毒就属于这种关系。
但是,病毒会变异,有些变异会导致病毒的宿主改变。病毒与新的宿主没有长期磨合的默契,就会出现“下手没轻没重”的问题,其中有些下手特别重的就会给宿主带来致命疾病。
在长期的演化中,人类已经和那些从远古祖先开始一路陪伴的病毒达成完美默契了,与从家畜那里来的病毒,比如麻疹、流感等等,磨合得还不完美,但多少有点默契,所以极少引起很严重的疫情。唯独来自于野生动物的病毒跟我们丝毫没有一丁点磨合,因此引起大瘟疫的疾病几乎全部来自野生动物。
因此希望大家平时尽量远离野生动物,包括流浪动物,更不要去饲养、食用野生动物,不然这些野生动物身上的病毒可就保不准拿你来试刀子啦。
愿小伙伴们都能用知识武装起自己,加油!
参考资料:
1.Wessner, D. R. (2010). The origins of viruses. Nature Education, 3(9), 37.
2.Suchard, M. A., Lemey, P., Baele, G., Ayres, D. L., Drummond, A. J., & Rambaut, A. (2018). Bayesian phylogenetic and phylodynamic data integration using BEAST 1.10. Virus evolution, 4(1), vey016.
3.Duffy, S. (2018). Why are RNA virus mutation rates so damn high?. PLoS biology, 16(8), e3000003.
4.Stern, A., Te Yeh, M., Zinger, T., Smith, M., Wright, C., Ling, G., ... & Andino, R. (2017). The evolutionary pathway to virulence of an RNA virus. Cell, 169(1), 35-46.
5.Villarreal, L. P., & DeFilippis, V. R. (2000). A hypothesis for DNA viruses as the origin of eukaryotic replication proteins. Journal of Virology, 74(15), 7079-7084.
6.Koonin, E. V., & Martin, W. (2005). On the origin of genomes and cells within inorganic compartments. TRENDS in Genetics, 21(12), 647-654.
7.Kolakofsky, D. (2015). A short biased history of RNA viruses. RNA, 21(4), 667-669.
8.Van Etten, J. L., Lane, L. C., & Dunigan, D. D. (2010). DNA viruses: the really big ones (giruses). Annual review of microbiology, 64, 83-99.
9.Prangishvili, D., Forterre, P., & Garrett, R. A. (2006). Viruses of the Archaea: a unifying view. Nature Reviews Microbiology, 4(11), 837-848.
10.Canchaya, C., Fournous, G., Chibani-Chennoufi, S., Dillmann, M. L., & Brüssow, H. (2003). Phage as agents of lateral gene transfer. Current opinion in microbiology, 6(4), 417-424.
11.Gerstein, M., & Zheng, D. (2006). The real life of pseudogenes. Scientific American, 295(2), 48-55.
12.Chuong, E. B., Elde, N. C., & Feschotte, C. (2017). Regulatory activities of transposable elements: from conflicts to benefits. Nature Reviews Genetics, 18(2), 71.
13.Sanmiguel, P., & Bennetzen, J. L. (1998). Evidence that a recent increase in maize genome size was caused by the massive amplification of intergene retrotransposons. Annals of Botany, 82, 37-44.
14.Bell, P. J. L. (2001). Viral eukaryogenesis: was the ancestor of the nucleus a complex DNA virus?. Journal of Molecular Evolution, 53(3), 251-256.
15.黄耀伟, 李龙, & 于涟. (2004). 人类及动物 RNA 病毒的反向遗传系统 (Doctoral dissertation).
16.Villarreal, L. P., & Villareal, L. P. (1997). On viruses, sex, and motherhood. Journal of Virology, 71(2), 859.
17.Dupressoir, A., Lavialle, C., & Heidmann, T. (2012). From ancestral infectious retroviruses to bona fide cellular genes: role of the captured syncytins in placentation. Placenta, 33(9), 663-671.
18.La Scola, B., Audic, S., Robert, C., Jungang, L., de Lamballerie, X., Drancourt, M., ... & Raoult, D. (2003). A giant virus in amoebae. Science, 299(5615), 2033-2033.
19.Xiao, C., Chipman, P. R., Battisti, A. J., Bowman, V. D., Renesto, P., Raoult, D., & Rossmann, M. G. (2005). Cryo-electron microscopy of the giant Mimivirus. Journal of molecular biology, 353(3), 493-496.
20.Schulz, F., Roux, S., Paez-Espino, D., Jungbluth, S., Walsh, D., Denef, V. J., ... & Woyke, T. (2020). Giant virus diversity and host interactions through global metagenomics. Nature, 1-7.
21.Brahim Belhaouari, D., Baudoin, J. P., Gnankou, F., Di Pinto, F., Colson, P., Aherfi, S., & La Scola, B. (2019). Evidence of a Cellulosic Layer in Pandoravirus massiliensis Tegument and the Mystery of the Genetic Support of Its Biosynthesis. Frontiers in Microbiology, 10, 2932.
22.Forterre, P. (2010). Giant viruses: conflicts in revisiting the virus concept. Intervirology, 53(5), 362-378.
23.Shabbir, M. Z., Rahman, A. U., & Munir, M. (2020). A comprehensive global perspective on phylogenomics and evolutionary dynamics of Small ruminant morbillivirus. Scientific Reports, 10(1), 1-17.
本文仅代表作者观点,不代表中国科普博览立场


 朗读本文
朗读本文




























 渝公网安备50010702502703号
渝公网安备50010702502703号